Im Projekt EDIT wurde in Kooperation mehrerer Projektpartner ein innovatives Monitoring-System zum schnellen Nachweis von Viren und Bakterien in Trink- und Rohwasser entwickelt.
Für Trinkwasserversorger ist neben der Sicherstellung hoher Trinkwasserqualität auch die qualitative Beurteilung des genutzten Rohwassers von großem Interesse. In der Praxis wird die mikrobiologische Wasserqualität auf Basis von Indikatorbakterien bewertet. Eine vollständige Überwachung auf alle bekannten Mikroorganismen ist in der Praxis aufgrund des extrem hohen Aufwands kaum möglich.
Indikatorbakterien werden in Kulturverfahren durch deren Wachstum in Nährmedien nachgewiesen. Dieser Nachweis benötigt bei sich schnell vermehrenden Bakterien wie E. coli 18 Stunden und kann vor allem bei Viren bis zu zwei Wochen dauern.
Ein schneller Nachweis aller hygienisch relevanter Organismen ist mit molekularbiologischen Nachweisverfahren möglich. Diese sind in der medizinischen Diagnostik bereits seit Jahren etabliert, eigenen sich jedoch durch ihre geringe Empfindlichkeit nicht für einen direkten Einsatz im Trinkwasserbereich.
Im BMBF-Projekt EDIT wurde ein mehrstufiges Verfahren zum schnellen molekularbiologischen Nachweis von Viren und Bakterien entwickelt und getestet.
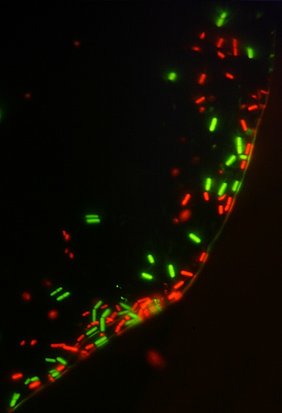
Das TZW übernahm im Projekt, neben der Validierung des Detektionssystems, die Entwicklung der Lebend/tot-Differenzierung. Wird ein Bakterium oder ein Virus inaktiviert, kann es dennoch mit molekularbiologischen Methoden erfasst werden. Für hygienische Bewertungen und Risikoeinschätzungen sind jedoch nur die Organismen von Belang, die eine Infektion auslösen können.
Mit Hilfe von fluoreszenten Farbstoffen konnten in Laborversuchen tote Bakterien und inaktivierte Viren vom molekularbiologischen Nachweis ausgeschlossen werden. Im Rahmen des Projektes wurden außerdem verschiedene Desinfektionsverfahren wie Ozon, Chlor und UV-Strahlung untersucht und mit dem Verfahren getestet.
Veröffentlichungen:
Elsäßer D., Ho J., Niessner R., Tiehm A., Seidel M.: Heterogeneous asymmetric recombinase polymerase amplification (haRPA) for rapid hygiene control of large-volume water samples. Analytical Biochemistry 546: 58-64 (2018) DOI 10.1016/j.ab.2018.01.032
Ho J., Seidel M., Niessner R., Eggert J., Tiehm A.: Long amplicon (LA)-qPCR for the discrimination of infectious and noninfectious phiX174 bacteriophages after UV inactivation. Water Research 103: 141-148 (2016) DOI 10.1016/j.watres.2016.07.032
Karthe D., Behrmann O., Blättel V., Elsässer D., Heese C., Hügle M., Hufert F., Kunze A., Niessner R., Ho J., Scharow B., Spoo M.,Tiehm A., Urban G., Vosseler S., Westerhoff T., Dame G., Seidel M.: Modular development of an inline monitoring system for waterborne pathogens in raw and drinking water. Environmental Earth Sciences 75: 1481 (2016) DOI 10.1007/s12665-016-6287-9
Lengger S., Otto J., Elsässer D., Schneider O., Tiehm A., Fleischer J., Niessner R., Seidel M.: Oligonucleotide microarray chip for the quantification of MS2, ϕX174 and adenoviruses on the multiplex analysis platform MCR3. Analytical and bioanalytical chemistry, 406(14), 3323-3334 (2014) Doi: 10.1007/s00216-014-7641-y
Ho, J.: Molekularbiologische lebend/tot-Unterscheidung. Veröffentlichungen aus dem Technologiezentrum Wasser, ISSN 1434-5765, TZW-Band 81 (2017)
Die Bände der TZW-Schriftenreihe können hier bestellt werden.