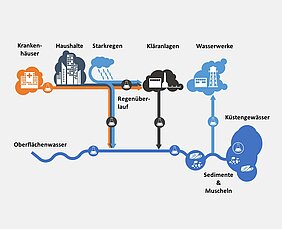
Ein besseres Verständnis des Verbleibs pathogener Viren und antibiotikaresistenter Bakterien von den Quellen über die Flusseinzugsgebiete bis zu den Mündungen ist dringend erforderlich. Das internationale Forschungsprojekt SARA unter der Koordination des TZW konnte einige Wissenslücken schließen.
Im Rahmen dieses Projekts wurde die Prävalenz pathogener Viren (einschließlich SARS-CoV-2), mikrobieller Indikatoren, Antibiotikaresistenzen und Microbial Source Tracking (MST)-Marker in Abwasser, Oberflächenwasser, Küstengewässern, Sedimenten und Muscheln in Einzugsgebieten in verschiedenen Klimazonen (Schweden, Deutschland, Frankreich, Spanien, Portugal, Israel, Mosambik und Uganda) bestimmt. Die Ziele des Projekts waren: (i) Harmonisierung der Methoden und Schulung der europäischen und afrikanischen Partner, (ii) Nachweis von SARS-CoV-2 in Rohabwasser als Biomarker für COVID-19-Fälle, (iii) Überwachung von enteralen Viren, Antibiotikaresistenzen und MST-Markern in der aquatischen Umwelt, (iv) Bewertung von Sedimenten und Muscheln als integrale Elemente, (v) Bestimmung der Auswirkungen von Extremwetterereignissen und (vi) mikrobielle Risikobewertung mit den gewonnenen Daten. Das Projekt wurde von einem Stakeholder Forum begleitet, dass sich aus Vertretern internationaler Gesundheits- und Umweltorganisationen sowie Behörden und Wasserwerken zusammensetzte.
Für die Untersuchungen wurden sowohl Kulturmethoden als auch molekularbiologische und metagenomische Methoden eingesetzt. Die kulturbasierten Analysen wurden in allen Partnerlaboratorien innerhalb von 24 bzw. 48 Stunden durchgeführt. Für die molekularen Analysen wurden die zeitnah angereichert, extrahiert und die resultierenden Nukleinsäureextrakte bis zum Versand an das spezialisierte Partnerlabor bei -80°C gelagert. Um die Vergleichbarkeit der Ergebnisse zu gewährleisten, wurden diese Verfahren in der ersten Projektphase harmonisiert und in einer Broschüre zusammengefasst (verfügbar über die SARA-Projekthomepage).
Besonderes Augenmerk wurde auf den Aufbau von Kapazitäten bei den afrikanischen Partnern gelegt. Nach der erfolgreichen Harmonisierung der Methoden halfen die europäischen Partner bei der Einrichtung der afrikanischen Partnerlabore und führten ein umfassendes Training für das afrikanische Laborpersonal durch. Das Training umfasste die harmonisierten Methoden zum kulturellen Nachweis von Indikatormikroorganismen und antibiotikaresistenten Bakterien sowie die ersten Konzentrations- und Extraktionsschritte der Proben für die molekularbiologische Analyse.
Die Projektergebnisse zeigen:
- Vorteile der abwasserbasierten Überwachung auf SARS-CoV-2
- Unterschiede in der Präsenz der untersuchten humanpathogenen Viren zwischen den verschiedenen Einzugsgebieten und Kläranlagen
- Unterschiede in der Prävalenz und relativen Häufigkeit von Extended Spectrum Beta-Lactamase (ESBL)-produzierenden E. coli und Antibiotikaresistenzgenen zwischen den Modellstandorten, mit der höchsten Abundanz in Uganda und der geringsten Abundanz in Schweden
- eine höhere Anzahl an somatischen Coliphagen als virale Indikatoren im Vergleich zu F-spezifischen Bakteriophagen
- die begrenzte Reduktion aller untersuchten mikrobiologischen Parameter durch die einfache Abwasserbehandlung in Uganda (Klärteiche) in ihrer derzeitigen Betriebsweise
- Zunahme der mikrobiellen Verunreinigung nach extremen Ereignissen (starke Regenfälle)
- hohe Persistenz von antibiotikaresistenten oligotrophen Umweltbakterien als wichtiges Reservoir für Antibiotikaresistenzen in der Umwelt
- Auftreten von allen drei erfassten humanen MST-Markern und damit den hohen Anteil menschlicher fäkaler Verunreinigungen an allen Modellstandorten
- Zunehmende Hinweise darauf, dass Bakteriophagen eine wichtige Rolle bei der Verbreitung von Antibiotikaresistenzgenen in aquatischen Ökosystemen spielen.
Die wichtigsten Erkenntnisse aus dem Vorhaben wurden in einem Dokument zusammengefasst, welches auf der
SARA-Projekthomepage eingesehen werden kann.
Veröffentlichungen
Abaasa C. N., Stange C., Ayesiga S., Mulogo E. M., Rogers K., Lejju J. B., Andama M., Tamwesigire I. K., Bazira J., Byarugaba F., Tiehm A. Antibiotic resistance of E. coli isolates form different water sources in Mbarara, Uganda. J. Water Health (2024) DOI: 10.2166/wh.2024.319
Holzer, C.; Ho, J.; Tiehm, A.; Stange, C.: Wastewater monitoring - passive sampling for the detection of SARS-CoV-2 and antibiotic resistance genes in wastewater. The Science of the Total Environment 959, 178244 (2025) DOI: 10.1016/j.scitotenv.2024.178244
Stange C.,Sidhu J.P.S.,Tiehm A.,Toze S.: Antibiotic resistance and virulence genes in coliform water isolates. International Journal of Hygiene and Environmental Health 219: 823-831 (2016) DOI.org/10.1016/j.ijheh.2016.07.015
Stange, C.; Yin, D.; Xu, T.; Guo, X.; Schäfer, C.; Tiehm, A.: Distribution of clinically relevant antibiotic resistance genes in Lake Tai, China. Science of the total environment 655: 337–346 (2019) DOI: 10.1016/j.scitotenv.2018.11.211
Ho J., Seidel M., Niessner R., Eggert J., Tiehm A.: Long amplicon (LA)-qPCR for the discrimination of infectious and noninfectious phiX174 bacteriophages after UV inactivation. Water Research 103: 141-148 (2016) DOI 10.1016/j.watres.2016.07.032
Poster
Stange C., Ho J., Sanchez-Cid C., Mulogo E., Nasser A., Nhantumbo C., Monteiro S., Simonsson M., Blanch A.R., Vogel T.M, Tiehm A.: Wastewater-based epidemiology for monitoring COVID-19 and antimicrobial resistance: Insights from comprehensive studies. International Conference Towards a Global Wastewater Surveillance System for Public Health, 15-17 Nov 2023, Frankfurt am Main, Deutschland (pdf-Datei)