Die besorgniserregende Zunahme von Antibiotikaresistenzen bezeichnet die Weltgesundheitsorganisation WHO als „stille Pandemie“. Die langjährige und umfangreiche Verwendung von Antibiotika hat die Bildung und Verbreitung von antibiotikaresistenten Bakterien und Antibiotikaresistenzgenen nicht nur im klinischen Bereich, sondern auch in der aquatischen Umwelt begünstigt. Aus diesem Grund werden antibiotikaresistente Bakterien und Antibiotikaresistenzgene als neue Parameter zur Beurteilung der hygienischen Wasserqualität zunehmend diskutiert. Das TZW verfügt durch eine Reihe von abgeschlossenen und laufenden Forschungsvorhaben über langjährige Erfahrungen zum Nachweis von Antibiotikaresistenzen in der Umwelt. In diesem Beitrag stellen wir ein aktuell beendetes Projekt und seine Ergebnisse vor.
Im Rahmen eines vom DVGW über dreieinhalb Jahre geförderten Projektes wurden angepasste Methoden zum Nachweis von antibiotikaresistenten Bakterien in der aquatischen Umwelt und von Resistenzgenen nach der Trinkwasseraufbereitung und Desinfektion entwickelt und angewandt.
Nachweis von resistenten Umweltbakterien
Antibiotikaresistenzen und Spuren von Antibiotika gelangen über Abwässer oder die Landwirtschaft, z. B. durch Ausbringung von Gülle, in die aquatische Umwelt. Der langjährige Eintrag hat die Verbreitung von Resistenzen unter Umweltbakterien gefördert, die an die nährstoffarmen Bedingungen von Oberflächengewässern angepasst sind. Von besonderer Relevanz sind dabei von der WHO als kritisch eingestufte Antibiotikaresistenzen gegen klinische Reserveantibiotika. Reserveantibiotika sind spezielle Antibiotika, die nur bei Infektionen mit Erregern angewandt werden, die gegen gängige Antibiotika bereits resistent sind. Im Rahmen aktueller Arbeiten wurden neue Kulturverfahren entwickelt, die ein spezifisches Monitoring resistenter Umweltbakterien ermöglichen. Es konnte gezeigt werden, dass nicht nur Resistenzen gegen häufig verabreichte Antibiotika, sondern auch gegen Reserveantibiotika unter aquatischen Umweltbakterien bereits weit verbreitet sind. Eine Reduktion der Einträge an den Quellen ist dringend erforderlich.
Nachweis von Resistenzgenen nach Aufbereitung und Desinfektion
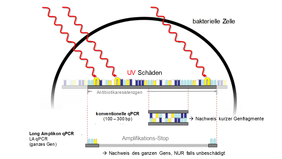
Reaktive Verfahren wie UV-Bestrahlung, Chlorung und Ozonung sind in der Trinkwasseraufbereitung gängige Techniken. Sie führen zu DNA-Schäden und können somit auch resistente Bakterien und Resistenzgene inaktivieren. Antibiotikaresistenzgene haben eine Größe von bis zu 2000 Basenpaaren und müssen als intaktes Gen vorliegen, um eine Resistenz zu vermitteln. Zum Nachweis von Resistenzgenen in der molekularbiologischen Standardmethodik (qPCR), werden nur kleine Abschnitte dieser Gene (100 bis 300 Basenpaare) untersucht. Sind genau diese Teilabschnitte nach reaktiven Behandlungen nicht geschädigt, kann es zu falsch-positiven Ergebnissen kommen. Um die Eliminationsleistungen realistisch zu erfassen, wurden erfolgreich sogenannte Long Amplikon Verfahren entwickelt, die die vollständigen Resistenzgene nachweisen. Es konnte in Versuchen mit Modell- und Realproben gezeigt werden, dass die Eliminationsleistung der drei reaktiven Verfahren mit der Standardmethode gegenüber der Long Amplikon Methode um bis zu Faktor 90 unterschätzt wird.
Fazit und Ausblick
Die Ergebnisse des Projektes bestätigen die Relevanz von Umweltbakterien als Reservoir für Antibiotikaresistenzen und bieten die Möglichkeit eines optimierten Nachweises nach einer reaktiven Wasserbehandlung. Der Nachweis und die Elimination von Antibiotikaresistenzen werden derzeit in Projekten zur weitergehenden Behandlung und Wiederverwendung von Abwasser („Nutzwasser“) sowie in einer internationalen Kooperation mit acht Partnern in Europa und Afrika weiterentwickelt und vergleichend untersucht („SARA – Surveillance of Emerging Pathogens and Antibiotic Resistances in Aquatic Ecosystems“).
Links
Projektseite EVA auf TZW-Website
Projektseite EVA auf DVGW-Website
Projektwebsite SARA
Veröffentlichungen
Stange C., Tiehm A.: Verhalten von Antibiotika-Resistenzgenen bei der Trinkwasseraufbereitung. Veröffentlichungen aus dem Technologiezentrum Wasser, ISSN 1434-5765, TZW-Band 76 (2017)
Stange C.,Sidhu J.P.S.,Tiehm A.,Toze S.: Antibiotic resistance and virulence genes in coliform water isolates. International Journal of Hygiene and Environmental Health 219: 823-831 (2016) DOI 10.1016/j.ijheh.2016.07.015
Stoll C., Sidhu J.P.S., Tiehm A., Toze S.: Prevalence of clinically relevant antibiotic resistance genes in surface water samples collected from Germany and Australia. Environmental Science & Technology 46: 9716-9726 (2012) DOI.org/10.1021/es302020s
Stange C., Sidhu J.P.S., Toze S., Tiehm A.: Comparative removal of antibiotic resistance genes during chlorination, ozonation, and UV treatment. Int. J. Hyg. Environ. Health 222: 541-548 (2019) DOI 10.1016/j.ijhe.2019.02.002
Stange C., Yin D., Xu T., Guo X., Schäfer C., Tiehm A.: Distribution of clinically relevant antibiotic resistance genes in Lake Tai, China. Science of the Total Environment 655: 337-346 (2019) DOI 10.1016/j.scitotenv.2018.11.211
Stange, C.; Tiehm, A. (2020): Occurrence of antibiotic resistance genes and microbial source tracking markers in the water of a karst spring in Germany. The Science of the total environment 742, S. 140529. DOI: 10.1016/j.scitotenv.2020.140529.